Un conjunto de artículos publicados en la revista Nature ofrecen la más completa caracterización y clasificación de tipos celulares del cerebro de este mamífero. Los resultados ayudan a comprender mejor la organización de este órgano, los circuitos neuronales y la función de cada una de sus células, además de contribuir a la investigación de los trastornos neurológicos en humanos.
Las complejas y diversas actividades que realizan los cerebros de los mamíferos, incluido el humano, están controladas por circuitos neuronales altamente especializados compuestos por numerosos tipos celulares con propiedades variables. Para entender cómo funciona este complejo órgano se necesita conocer en detalle la organización y función de esta variedad de células y los circuitos cerebrales.
Hasta este año, los intentos para explorar el cerebro se han limitado a las diferentes regiones seleccionadas, pero la Red del Censo Celular de la Iniciativa BRAIN (BICCN, por sus siglas en inglés) ha publicado recientemente el primer borrador del mapa de las células del cerebro humano y ahora, en la revista Nature, nueve artículos que ofrecen un pormenorizado análisis del cerebro del ratón con su diversidad celular.
El paquete de artículos presenta la caracterización y clasificación más completa y detallada de tipos celulares de este órgano del roedor realizada hasta la fecha. Los resultados permiten comprender mejor la estructura y organización del cerebro.
El trabajo proporciona una herramienta para continuar las investigaciones sobre el desarrollo y la evolución del cerebro de los mamíferos, incluido el modo en que la organización de los distintos tipos celulares puede contribuir a los trastornos neurológicos que afectan a los humanos.
En uno de los artículos, los investigadores Zizhen Yao y Hongkui Zeng del Instituto Allen de Ciencias del Cerebro (EE UU) y sus colegas describen cómo han creado el mapa cerebral de alta resolución, a partir de una combinación de secuenciación de ARN unicelular de unos 4 millones de células y datos transcriptómicos [transcriptos de ARN o copias de la secuencia de ADN de un gen] espaciales de unos 4,3 millones de células.
Esta transcriptómica espacial, que permite posicionar dentro del mapa cerebral los distintos tipos celulares, es una de la novedades del trabajo, un avance que ha sido posible gracias a nuevas técnicas de microscopía aplicadas sobre los cortes histológicos efectuados al cerebro de ratón.
El atlas de los tipos celulares se presenta en cuatro niveles de clasificación organizados jerárquicamente: 34 clases, 338 subclases, 1.201 supertipos y 5.322 grupos o clusters.
Los resultados revelan características únicas de la organización de la diversidad celular en las distintas regiones cerebrales: la parte dorsal contiene pocos tipos celulares pero muy diversos, mientras que la parte ventral contiene tipos neuronales más numerosos y más estrechamente relacionados entre sí.
También mediante la transcriptómica espacial y una técnica llamada Slide-seq (permite posicionar los transcritos de ARN usando códigos de barras de ADN), otro equipo liderado por los investigadores Evan Macosko y Fei Chen del Instituto Broad (del MIT y Harvard) descubrieron aproximadamente el 90 % de todas las poblaciones celulares del cerebro de ratón, revelando la sorprendente diversidad celular en regiones cerebrales poco estudiadas, especialmente el mesencéfalo, la protuberancia, la médula y el hipotálamo.
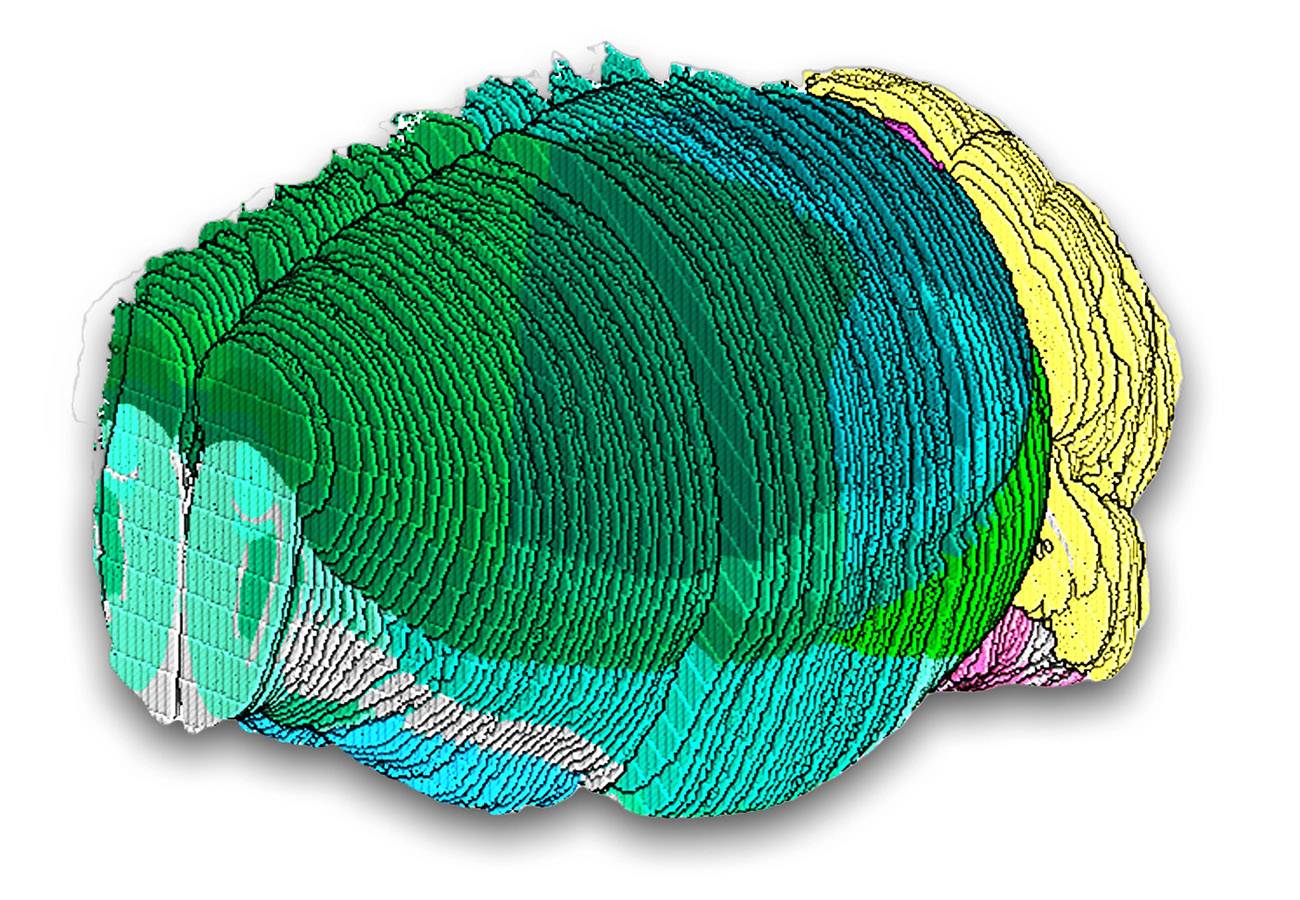
Los investigadores analizaron 101 cortes en el cerebro de un ratón, brindándoles una visión profunda y completa de los tipos de células en todo el cerebro (con sus regiones coloreadas). / Macosko and Chen labs
"Sospechábamos que la mayor diversidad se encontraría en estas regiones, así que les dimos prioridad en nuestro perfil", explica Macosko, "y gran parte de lo que realmente hace el cerebro se encuentra en estas zonas básicas, que han recibido muy poca atención en comparación con el córtex; nuestros resultados subrayan la necesidad de estudiarlas más a fondo".
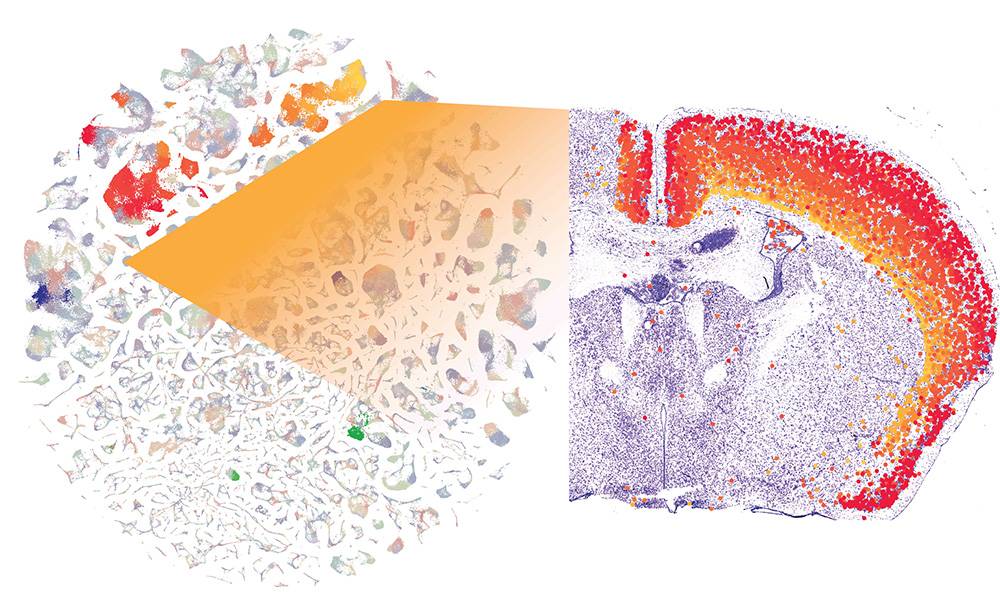
Ejemplo de tipos celulares del cerebro de ratón y su localización. / Chen and Macosko labs
En otro artículo, Joseph Ecker del Instituto Salk de Estudios Biológicos, Bing Ren de la Universidad de California en San Diego y otros autores comparan la regulación génica en una región cerebral, la corteza motora primaria, de humanos, macacos, titíes y ratones.
De esta forma han hallado características conservadas en estos cuatro tipos de mamíferos sobre variantes genéticas relacionadas con la esclerosis múltiple, la anorexia nerviosa y la adicción al tabaco. Estos hallazgos demuestran el valor de los mapas cerebrales para identificar variantes genéticas que contribuyen a enfermedades y rasgos neurológicos.
"Este trabajo nos está ayudando a establecer una comprensión de referencia de cómo es el cerebro a nivel celular", apunta Ren, "esto permitirá establecer comparaciones entre nuestra línea de base y cerebros con trastornos neurológicos y psiquiátricos. Estudiar el cerebro de este modo podría ayudarnos a descubrir nuevos enfoques terapéuticos para estas afecciones".
Por su parte, Ecker, destaca la importancia de conocer el cerebro de otros mamíferos: "El ser humano ha evolucionado a lo largo de millones de años, y gran parte de esa historia evolutiva la comparte con otros animales. Los datos de los humanos por sí solos nunca serán suficientes para decirnos todo lo que queremos saber sobre el funcionamiento del cerebro. Al rellenar estas lagunas con otras especies de mamíferos, podemos seguir respondiendo a esas preguntas y mejorar los modelos de aprendizaje automático que utilizamos proporcionándoles más datos".
Otros artículos identifican características genéticas específicas de cada célula que influyen en funciones especializadas, o exploran cómo los tipos celulares de distintas partes del cerebro forman conexiones o han ido evolucionando en diferentes especies.
Uno de los estudios, por ejemplo, se centra en los tipos celulares asociados a la retina del ojo, y los resultados revelan un origen común en los vertebrados, a pesar de que varíen mucho entre los diferentes grupos. Los peces necesitan ver bajo el agua, los ratones y los gatos requieren una buena visión nocturna, y los monos y los humanos desarrollaron una vista diurna muy aguda para cazar y buscar comida. Algunos animales ven colores vivos, mientras que otros se conforman con ver el mundo en blanco y negro.
Sin embargo, numerosos tipos de células son comunes a toda una serie de especies de vertebrados, lo que sugiere que los programas de expresión génica que definen estos tipos se remontan probablemente al ancestro común de los vertebrados con mandíbula, concluyen estos autores, liderados por Karthik Shekhar desde la Universidad de California en Berkeley (EE UU).
Además, el nuevo mapa detallado de los tipos celulares de retinas de vertebrados también podría ayudar a investigar enfermedades oculares humanas, como el glaucoma.
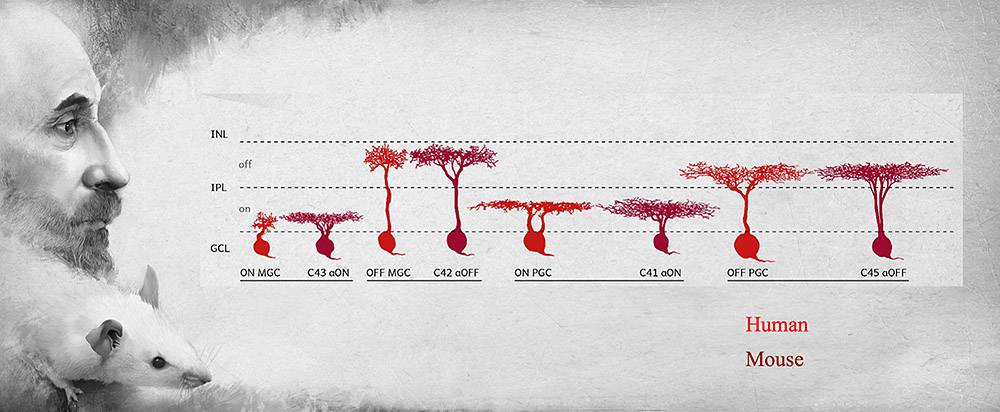
Comparación de células ganglionares de la retina humana y de ratón. / Hugo Salais, Metazoa Studio
"Los atlas de tipos de células son fundamentales no solo para comprender la arquitectura del cerebro a escala celular, sino también para hacer inferencias precisas sobre la evolución cerebral", valora la investigadora Maria Antonietta Tosches de la Universidad de Columbia (EE UU) en un artículo paralelo escrito junto a Heather Lee de la Universidad de Newcastle (Australia), quien concluye: "Los trabajos presentados en este número han sentado bases firmes para muchos descubrimientos importantes en neurobiología y trastornos neurológicos".
Referencias:
Hongkui Zeng et al. “A high-resolution transcriptomic and spatial atlas of cell types in the whole mouse brain”
Evan Macosko et al. “The molecular cytoarchitecture of the adult mouse brain”
Xiaowei Zhuang et al. “Molecularly defined and spatially resolved cell atlas of the whole mouse brain”
Edward Callaway et al. “Brain-wide Correspondence of Neuronal Epigenomics and Distant Projection”
Joseph Ecker et al. “Single-cell DNA methylome and 3D multi-omic atlas of the adult mouse brain”
Bing Ren et al. “Single-cell analysis of chromatin accessibility in the adult mouse brain”
Bing Ren et al. “Conserved and divergent gene regulatory programs of the mammalian neocortex”
Zhigang He et al. “A transcriptomic taxonomy of mouse brain-wide spinal projecting neurons”
Karthik Shekhar et al. “Evolution of neuronal cell classes and types in the vertebrate retina”
Nature, 2023